KEEPA
KI-basierte Entwicklung optimierter Endolysine und deren Produktion als Antibiotikaersatz im Tiersektor
Ziel ist die Entwicklung eines proteinbasierten Antibiotikums/Desinfektionsmittels gegen Clostridium perfringens durch Einsatz phagenderivierter Endolysine. Mittels KI-gestütztem Proteindesign und optimierter Pichia pastoris-Expression werden Stabilität, Ausbeute und Wirksamkeit verbessert.
Forschungsgebiet:
- Laufzeit:
- 01.01.2026 - 31.12.2029
- Projektstatus:
- laufend
- Einrichtungen:
- Fakultät für Wirtschaftsingenieurwesen
- Projektleitung:
- Prof. Dr. Robert Huber, Prof. Dr. David Kostner
- Förderprogramm:
- Forschungsvorhaben von Hochschulen für Angewandte Wissenschaften in Kooperation mit Praxispartnern (HAW-ForschungsPraxis)
- Drittmittelart:
- Bund
- Projektart:
- Forschung
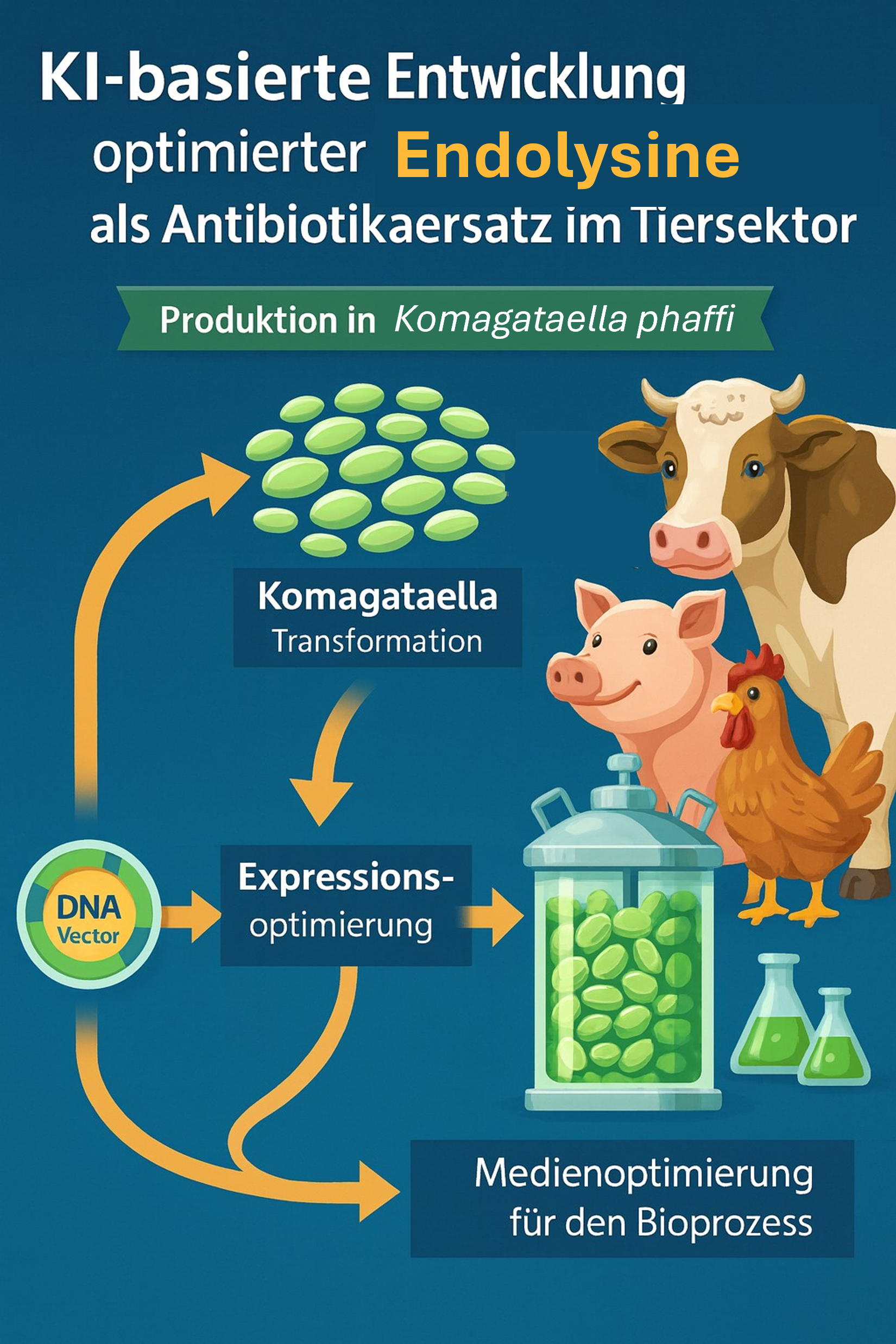
Die Verbundpartner beabsichtigen die Entwicklung eines umweltfreundlichen, proteinbasierten Desinfektionsmittelkandidaten zur Bekämpfung von Clostridium perfringens in der Tierhaltung.
Zu diesem Zweck nutzen die Partner hoch selektive Endolysine, bakterienzellwandlysierende Enzyme aus Bakterienviren, den sogenannten Bakteriophagen. Der Projektvorschlag der Hochschulen Bremen und München knüpft an vergangene Arbeiten der Praxispartner S2B GmbH & Co. KG, KTM GmbH und NOVAgreen GmbH an, in welchen die Machbarkeit der Herstellung von Endolysinen in der Hefe Pichia pastoris erfolgreich demonstriert und deren Anwendungspotential als Desinfektionsmittel gezeigt werden konnte. Für eine wirtschaftlich tragbare Anwendung ist jedoch eine signifikante Effizienzsteigerung der Proteinherstellung sowie eine Optimierung der Produktstabilität erforderlich. Um diese Zielstellungen zu erreichen, nutzen die Projektpartner einen mehrstufigen Optimierungsansatz auf fünf methodischen Ebenen und bedienen sich dazu neuester Erkenntnisse aus Wissenschaft und Forschung. Die Hochschule Bremen (HSB) optimiert in einem ersten Schritt die Temperatur- und pH-Stabilität von zwei C.perfringens – Endolysinen und nutzt dazu KI-unterstütze Proteindesignwerkzeuge sowie ein Expressionsscreening im Hochdurchsatzformat. Die dabei erzielten Verbesserungen in der Stabilität und Expressionsausbeute der Zielproteine werden mittels Differenzieller Scanning Fluorimetrie (DSF) und Dynamischer Lichtstreuung (DLS) erfasst. In einem zweiten Schritt erfolgt ein Transfer der Erkenntnisse in den Produktionsorganimus P. pastoris. In diesem Schritt arbeiten die Hochschule München (HM) und HSB gemeinsam an der Verbesserung der Proteinexpression und Sekretionsleistung. Die HM exprimiert zunächst Wildtypendolysine in unterschiedlichen, sekretionsoptimierten P.pastoris Hintergrundstämmen und nutzt dazu neben den klassischen integrativen Expressionsplasmiden ein konstitutives, episomales Vektorsystem mit erhöhter Kopienzahl. Eine vergleichende Quantifizierung der erzielten Endolysintiter soll eine konkrete Auswahl des optimalen genetischen Hintergrundes erlauben. Im späteren Projektverlauf nutzt die HSB diese Erkenntnisse um ein neuartiges, stabilitätsoptimiertes Derivat der ursprünglichen Endolysine mittels der gewählten sekretionsoptimierten P.pastoris Hintergrundstämme herzustellen. Dabei wird das endolysinkodierende Gen mittels eines Deep Learning Algorithmus für P.pastoris in 10 Variationen codonoptimiert und getestet. In einem dritten Schritt wird nun ein aus der Stammentwicklung hervorgegangener P.pastoris - Expressionsstamm in die Bioprozessentwicklung überführt. Die HM bedient sich dazu verschiedener Hochdurchsatz-Kultivierungssysteme wie Mikrotiterplatten, Schüttelkolben und automatisierten Mikrobioreaktorsystemen mit Online-Monitoring sowie einer neuartigen Methodik zur Messung der Sauerstofftransferrate in 96-well Mikrotiterplatten. Zusammen mit dem Praxispartner Labmaite GmbH wird zudem eine Machine Learning-gestützte Software zur reiterativen Nährmedien- und Expressionsverbesserung für die Endolysinherstellung weiterentwickelt. Während die HM darauf aufbauend einen Fed-Batch Bioprozess im Kleinst- und Labormaßstab entwickelt, übernimmt der Praxispartner S2B die spätere Skalierung des Upstreamprozesses in den Pilotmaßstab sowie die Formulierung der Endolysinpräparationen als Desinfektionsmittel. Um die spätere industrielle Reinigung der Endolysine technisch zu vereinfachen und die Herstellungskosten damit signifikant zu senken, versucht die HSB in einem 4. Schritt sich die neu gewonnene Temperaturstabilität der Endolysine zunutze zu machen, um mittels eines Hochtemperaturschrittes störende Proteinkontaminationen des P. pastoris Sekretoms abzureichern und die Zielproteine mittels Tangentialflußfiltration zu reinigen. Eine kontinuierliche Überprüfung der enzymatischen Aktivität sowie eine finale Evaluierung der Wirksamkeit der hergestellten Endolysine in Anwendungsumgebung erfolgt in Kooperation zwischen HSB und dem Praxispartner KTM.
Ausdrückliche Zielsetzung des Projektverbundes ist es, einen unmittelbaren Transfer von stabilen Endolysinkandidaten als zukünftige Antibiotika- / Desinfektionsersatzstoffe in die Anwendung zu beschleunigen.
Projektförderung

Projektträger
Adressierte Nachhaltigkeitsziele (SDGs)
